Fettleber ist nicht gleich Fettleber: Ein Team aus ForscherInnen der Karl-Franzens-Universität Graz, der Medizinischen Universität Graz und der TU Graz zeigte erstmals, dass sich anhand der spezifischen Zusammensetzung von Fett-Tröpfchen der Leberzellen auf die unterschiedlichen Ursachen der Krankheit schließen lässt. Dieses Wissen ist der Grundstein für die Entwicklung neuer Therapiemethoden. Die Forschungsergebnisse wurden im renommierten Journal of Lipid Research publiziert – und eroberten die Titelseite.
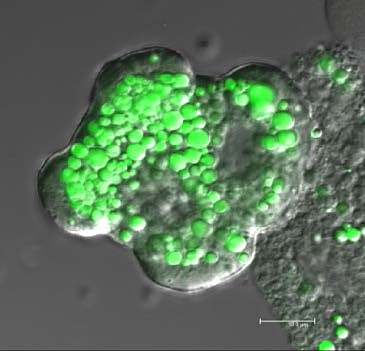
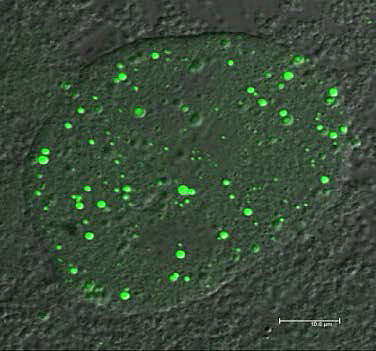
Fettes Essen, Fasten oder genetische Vorbelastung: Eine Fettleber kann aus unterschiedlichen Stoffwechselstörungen entstehen. So wird die „Nichtalkoholische Fettleber“ durch übermäßig fettes Essen erworben, in Verbindung mit einer Insulin-Resistenz. Eine andere Ursache kann eine genetisch bedingte Blockade des fettspaltenden Enzyms ATGL sein. Auch Fasten führt zu einer vermehrten Fetteinlagerung in die Leber, da die Lipide, also Fette, aus anderen Stellen im Körper in die Leber wandern. Welche Stoffwechselstörung einer Fettleber zugrunde liegt, darüber gibt die Zusammensetzung der Lipid-Tröpfchen von Leberzellen Auskunft, wie Grazer WissenschafterInnen im Rahmen eines von der EU geförderten Projekts nun herausgefunden haben.
Charakteristisches Fett-Muster
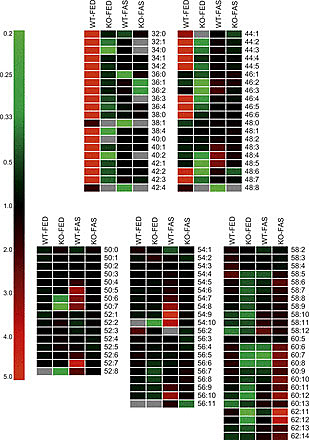
„Es zeigte sich, dass die Zusammensetzung der Lipid-Tröpfchen variiert“, berichtet Univ.-Prof. Dr. Friedrich Spener vom Institut für Molekulare Biowissenschaften der Karl-Franzens-Universität Graz. „Je nach Krankheitsursache sind in den Tröpfchen verschiedene Lipid-Moleküle in unterschiedlichen Mengen vorhanden. Daraus ergibt sich jeweils ein charakteristisches Muster, das erkennen lässt, ob die Fettleber Folge der verschiedenen Arten von Ernährungsstress oder genetischem Stress ist. Besonders geeignet zur Unterscheidung ist das Profil der Triglyzeride“, fasst der Biochemiker und Molekularbiologe zusammen.
Forschung im Grazer Trio
Die drei Grazer Universitäten, die unter der Flagge „BioTechMed-Graz“ ihre Forschung an der Schnittstelle zwischen Mensch, Medizin und Technik bündeln, haben auch in diesem Projekt ihre Kräfte vereint und Arbeitsteilung betrieben. Das Team um Friedrich Spener isolierte Lipid-Tröpfchen aus den Leberepithelzellen, die dann mittels Massenspektrometrie (MS) von Dr. Harald Köfeler und seinen MitarbeiterInnen am Zentrum für Medizinische Forschung der Med Uni Graz untersucht wurden. Dr. Gerhard Thallinger und sein Team vom Institut für Genomik und Bioinformatik der TU Graz haben die Ergebnisse mit eigens entwickelten bioinformatischen Verfahren ausgewertet. Die methodische Innovation liegt einerseits im Einsatz einer speziellen MS-Plattform, die im Hochdurchsatz-Verfahren große Mengen quantitativer Messdaten liefert, andererseits im bioinformatischen Auswerteverfahren, das die automatisierte Identifizierung der einzelnen Lipid-Moleküle und in ihrer Gesamtheit die Mustererkennung erlaubt. Dieser „lipidomische Ansatz“ ist die Methode der Wahl, denn insgesamt, so Schätzungen, gibt es über 100.000 verschiedene Lipid-Moleküle. „Diese Methode zur automatisierten Quantifizierung der Lipid-Moleküle ist zum Patent eingereicht. Es ist langfristig ein Ansatz, der auch in der klinischen Diagnostik eingesetzt werden kann“, erklärt der Bioinformatiker Gerhard Thallinger.
Graz als Zentrum europäischer Fettforschung
Graz ist neben Cambridge, Dresden, Lyon und Regensburg eines der großen Zentren der lipidomischen Forschung in Europa. Im Lipidomics Research Center Graz wurden auf Initiative von Friedrich Spener und Univ.-Prof. Dr. Sepp D. Kohlwein die Arbeitsgruppen am Standort unter einem virtuellen Dach zusammengeführt. Die Forschungen sind Teil der interuniversitären Plattform BioTechMed-Graz.
Originalpublikation: Chitraju, C., Trötzmüller M., Hartler J., Wolinski H., Thallinger G., Haemmerle G., Zechner R., Zimmermann R., Köfeler H., Spener F.: The impact of genetic stress by ATGL deficiency on the lipidome of lipid droplets from murine hepatocytes. Journal of Lipid Research. 2013, Vol. 54, Number 8, 2185-2194.